Pytorch와 TIAToolbox를 사용한 전체 슬라이드 이미지(Whole Slide Image) 분류#
번역: 박주환
팁
이 튜토리얼을 최대한 활용하려면, 이 Colab 버전 을 사용하는 것을 권장합니다. 이를 통해 아래의 자료를 실험해 볼 수 있습니다.
개요#
본 튜토리얼에서는 TIAToolbox를 사용한 PyTorch 모델을 통해 전체 슬라이드 이미지들(Whole Slide Images, WSIs)을 분류하는 방법을 알아보겠습니다. WSI란 수술이나 생검을 통해 채취된 인간 조직 샘플의 이미지이며, 이러한 이미지는 전문 스캐너를 이용해 스캔 됩니다. 이 데이터는 병리학자와 전산 병리학자들이 종양 성장에 대한 이해를 높이고 환자 치료를 개선하기 위해 암과 같은 질병을 미시적 수준에서 연구 하는데 사용됩니다.
WSIs를 처리하는 것이 어려운 이유는 엄청난 크기 때문입니다. 예컨대, 일반적인 슬라이드 이미지가 100,000x100,000 픽셀 정도의 크기를 가지며, 각 픽셀은 슬라이드 상에서 약 0.25x0.25 마이크로미터에 해당합니다. 이 때문에 이미지를 로드하고 처리하는 데 어려움을 야기하며, 한 연구에서 수백 개나 수천 개의 WSIs가 포함되는 경우는 말할 것도 없습니다 더 큰 연구가 더 나은 결과를 제공합니다!
전통적인 이미지 처리 파이프라인은 WSIs 처리에 적합하지 않으므로 더 나은 도구가 필요합니다. 이때, TIAToolbox 가 도움이 될 수 있는데, 이는 조직 슬라이드(tissue slides)를 빠르고 효율적으로 처리할 수 있는 유용한 도구들을 제공합니다. 일반적으로 WSIs는 시각화에 최적화된 다양한 배율에서 동일한 이미지의 여러 복사본이 피라미드 구조로 저장됩니다. 피라미드의 레벨 0(또는 가장 아래 단계)에는 가장 높은 배율 또는 줌 수준의 이미지를 포함하며, 피라미드의 상위 단계로 갈수록 기본 이미지의 저 해상도 사본이 있습니다. 하단에 피라미드의 구조가 그려져 있습니다.
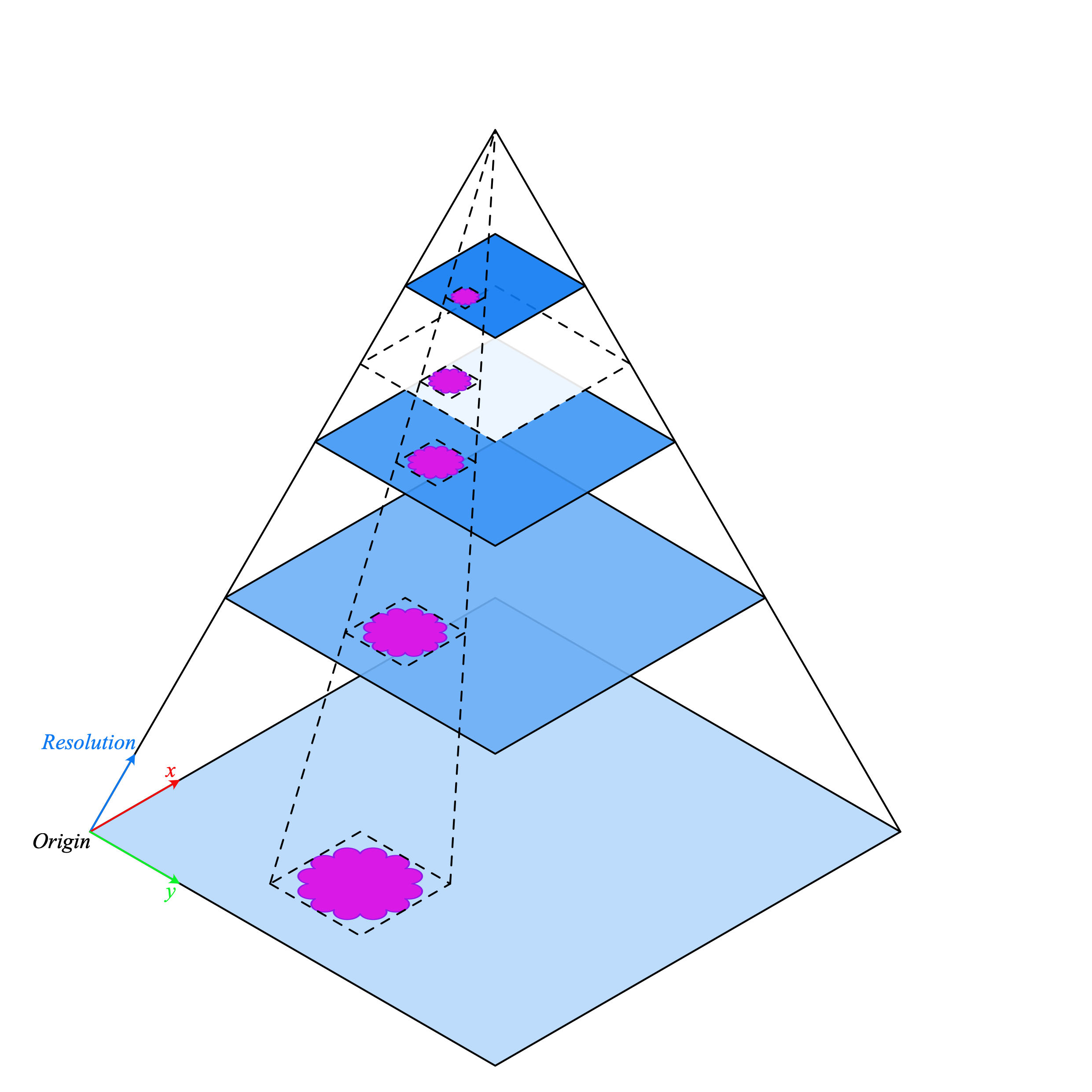 WSI 피라미드 stack
(source)
WSI 피라미드 stack
(source)
TIAToolbox를 사용하면 조직
분류 와 같은 일반적인
후속 분석 작업을 자동화할 수 있습니다. 본 튜토리얼에서는 다음을 수행하는
방법을 보여줍니다: 1. TIAToolbox를 사용하여 WSI(Whole Slide Image)를
이미지를 로드하는 방법 2. 서로 다른 Pytorch 모델을 사용하여 슬라이드를
패치 레벨(patch-level)에서 분류하는 방법. 본 튜토리얼에서는 TorchVision의
ResNet18 모델과 커스텀(custom) HistoEncoder
모델을 사용하는 예제를 제공합니다.
시작해보자!
환경설정#
본 튜토리얼에서 제공하는 예제를 실행하려면, 다음과 같은 패키지(packages)가 필요합니다.
OpenJpeg
OpenSlide
Pixman
TIAToolbox
HistoEncoder (for a custom model example)
이 패키지들을 설치하기 위해 터미널(terminal)에 아래의 명령어를 실행해주세요:
apt-get -y -qq install libopenjp2-7-dev libopenjp2-tools openslide-tools libpixman-1-dev
pip install -q ‘tiatoolbox<1.5’ histoencoder && echo “Installation is done.”
또한, MacOS에서는 apt-get 대신에 brew install openjpeg openslide
명령어를 실행하여 필수 패키지들을 설치할 수 있습니다.
설치에 대한 추가 정보는 여기
를 참조하세요.
관련 라이브러리 불러오기#
"""Jupyter 노트북을 실행하는 데 필요한 모듈 불러오기."""
from __future__ import annotations
# 로깅(logging) 설정하기
import logging
import warnings
if logging.getLogger().hasHandlers():
logging.getLogger().handlers.clear()
warnings.filterwarnings("ignore", message=".*The 'nopython' keyword.*")
# 데이터와 파일 다운로드하기
import shutil
from pathlib import Path
from zipfile import ZipFile
# 데이터 처리 및 시각화
import matplotlib as mpl
import matplotlib.pyplot as plt
import numpy as np
import pandas as pd
from matplotlib import cm
import PIL
import contextlib
import io
from sklearn.metrics import accuracy_score, confusion_matrix
# WSI 로딩 및 처리를 위한 TIAToolbox
from tiatoolbox import logger
from tiatoolbox.models.architecture import vanilla
from tiatoolbox.models.engine.patch_predictor import (
IOPatchPredictorConfig,
PatchPredictor,
)
from tiatoolbox.utils.misc import download_data, grab_files_from_dir
from tiatoolbox.utils.visualization import overlay_prediction_mask
from tiatoolbox.wsicore.wsireader import WSIReader
# Torch-관련
import torch
from torchvision import transforms
# 그래프 설정
mpl.rcParams["figure.dpi"] = 160 # for high resolution figure in notebook
mpl.rcParams["figure.facecolor"] = "white" # To make sure text is visible in dark mode
# 만약 GPU를 사용하지 않는다면, ON_GPU를 False로 변경하세요.
ON_GPU = True
# 장황한 코드 블록의 콘솔 출력을 억제하는 함수
def suppress_console_output():
return contextlib.redirect_stderr(io.StringIO())
실행 전 정리(clean-up)#
적절한 정리를 보장하기 위해(예컨대, 비정상 종료), 이번 실행에서
다운로드되거나 생성된 모든 파일은 global_save_dir 이라는
하나의 디렉토리에 저장되며, 이 디렉토리는 “./tmp/”로 설정됩니다.
유지보수를 쉽게 하기 위해 디렉토리 이름은 이 한 곳에서만 설정되므로,
필요하면 간편하게 변경할 수 있습니다.
warnings.filterwarnings("ignore")
global_save_dir = Path("./tmp/")
def rmdir(dir_path: str | Path) -> None:
"""디렉토리를 지우기 위한 도우미 함수"""
if Path(dir_path).is_dir():
shutil.rmtree(dir_path)
logger.info("Removing directory %s", dir_path)
rmdir(global_save_dir) # 이전 실행에서 디렉토리가 있는 경우 삭제
global_save_dir.mkdir()
logger.info("Creating new directory %s", global_save_dir)
데이터 다운로드#
샘플 데이터로는 전체 슬라이드 이미지(whole-slide image)를 사용하고 Kather 100k 데이터셋의 검증(validation) 하위 집단(subset)에서 추출한 패치들을 사용할 것입니다.
wsi_path = global_save_dir / "sample_wsi.svs"
patches_path = global_save_dir / "kather100k-validation-sample.zip"
weights_path = global_save_dir / "resnet18-kather100k.pth"
logger.info("Download has started. Please wait...")
# 전체 슬라이드 이미지(whole-slide image) 샘플을 다운로드 하고 압축을 해제하기
download_data(
"https://tiatoolbox.dcs.warwick.ac.uk/sample_wsis/TCGA-3L-AA1B-01Z-00-DX1.8923A151-A690-40B7-9E5A-FCBEDFC2394F.svs",
wsi_path,
)
# Kather 100K 데이터셋을 훈련하기 위해 사용된 검증 세트(validation set) 샘플을 다운로드하고 압축을 해제하기
download_data(
"https://tiatoolbox.dcs.warwick.ac.uk/datasets/kather100k-validation-sample.zip",
patches_path,
)
with ZipFile(patches_path, "r") as zipfile:
zipfile.extractall(path=global_save_dir)
# ResNet18 아키텍처로 WSI(전체 슬라이드 이미지) 분류를 위해 사전 학습된 모델 가중치를 다운로드하기
download_data(
"https://tiatoolbox.dcs.warwick.ac.uk/models/pc/resnet18-kather100k.pth",
weights_path,
)
logger.info("Download is complete.")
데이터 읽기#
패치 목록과 해당되는 라벨 목록을 생성합니다.
예를 들어, label_list 의 첫 번째 라벨은
patch_list 의 첫 번째 이미지 패치의 클래스를 나타냅니다.
# 패치 데이터를 읽고 패치 목록과 해당 라벨 목록을 생성
dataset_path = global_save_dir / "kather100k-validation-sample"
# 데이터셋 경로 설정
image_ext = ".tif" # 각 이미지의 파일 확장자
# 라벨 ID와 클래스 이름 간의 매핑
label_dict = {
"BACK": 0, # Background (empty glass region) # 이 부분은 밑에서 자세히 설명
"NORM": 1, # Normal colon mucosa
"DEB": 2, # Debris
"TUM": 3, # Colorectal adenocarcinoma epithelium
"ADI": 4, # Adipose
"MUC": 5, # Mucus
"MUS": 6, # Smooth muscle
"STR": 7, # Cancer-associated stroma
"LYM": 8, # Lymphocytes
}
class_names = list(label_dict.keys())
class_labels = list(label_dict.values())
# 패치 목록 생성 및 파일 이름에서 라벨 추출하기
patch_list = []
label_list = []
for class_name, label in label_dict.items():
dataset_class_path = dataset_path / class_name
patch_list_single_class = grab_files_from_dir(
dataset_class_path,
file_types="*" + image_ext,
)
patch_list.extend(patch_list_single_class)
label_list.extend([label] * len(patch_list_single_class))
# 데이터셋 통계 표기
plt.bar(class_names, [label_list.count(label) for label in class_labels])
plt.xlabel("Patch types")
plt.ylabel("Number of patches")
# 클래스별 예시 개수 집계
for class_name, label in label_dict.items():
logger.info(
"Class ID: %d -- Class Name: %s -- Number of images: %d",
label,
class_name,
label_list.count(label),
)
# 전체 데이터셋 통계
logger.info("Total number of patches: %d", (len(patch_list)))
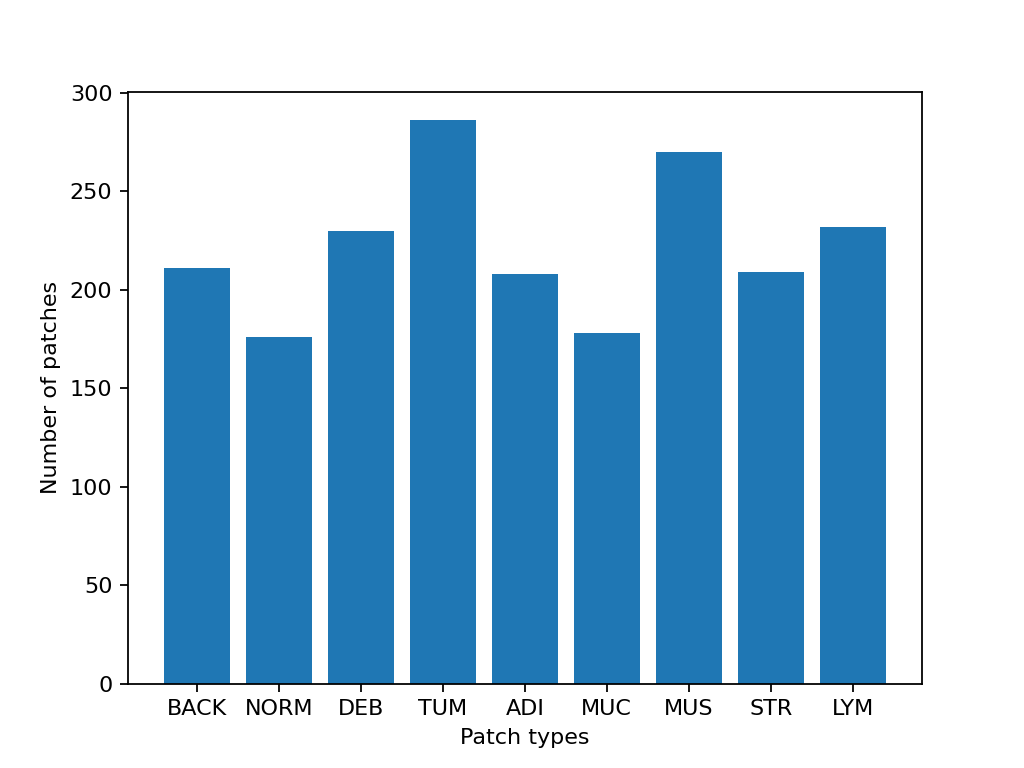
|2023-11-14|13:15:59.299| [INFO] Class ID: 0 -- Class Name: BACK -- Number of images: 211
|2023-11-14|13:15:59.299| [INFO] Class ID: 1 -- Class Name: NORM -- Number of images: 176
|2023-11-14|13:15:59.299| [INFO] Class ID: 2 -- Class Name: DEB -- Number of images: 230
|2023-11-14|13:15:59.299| [INFO] Class ID: 3 -- Class Name: TUM -- Number of images: 286
|2023-11-14|13:15:59.299| [INFO] Class ID: 4 -- Class Name: ADI -- Number of images: 208
|2023-11-14|13:15:59.299| [INFO] Class ID: 5 -- Class Name: MUC -- Number of images: 178
|2023-11-14|13:15:59.299| [INFO] Class ID: 6 -- Class Name: MUS -- Number of images: 270
|2023-11-14|13:15:59.299| [INFO] Class ID: 7 -- Class Name: STR -- Number of images: 209
|2023-11-14|13:15:59.299| [INFO] Class ID: 8 -- Class Name: LYM -- Number of images: 232
|2023-11-14|13:15:59.299| [INFO] Total number of patches: 2000
이 패치 데이터셋에서 볼 수 있듯이, 0부터 8까지의 ID를 가진 9개의 클래스와 라벨이 있으며, 각 클래스는 해당 패치에서 주로 나타나는 조직 유형을 설명합니다:
BACK ⟶ 배경(Background)(비어 있는 영역)
LYM ⟶ 림프구(Lymphocytes)
NORM ⟶ 정상 대장 점막(Normal colon mucosa)
DEB ⟶ 조직 파편(Debris)
MUS ⟶ 평활근(Smooth muscle)
STR ⟶ 암 관련 기질(Cancer-associated stroma)
ADI ⟶ 지방 조직(Adipose)
MUC ⟶ 점액(Mucus)
TUM ⟶ 대장선암 상(Colorectal adenocarcinoma epithelium)
이미지 패치 분류#
먼저 patch 모드를 사용하여 디지털 슬라이드 내의
각 패치에 대한 예측을 구하는 방법을 시연한 후, wsi 모드를 사용하여
큰(large) 슬라이드에 대해 예측을 수행하는 방법을 보여줍니다.
PatchPredictor 모델 정의하기#
PatchPredictor 클래스는 PyTorch로 작성된 CNN 기반 분류기를 실행합니다
모델은tiatoolbox.models.abc.ModelABC`(문서)<https://tia-toolbox.readthedocs.io/en/latest/_autosummary/tiatoolbox.models.models_abc.ModelABC.html>`__ 클래스 구조를 따르는 모든 PyTorch로 훈련된 모델을 사용할 수 있습니다. 이에 대한 자세한 내용은 고급 모델 기술에 관한 예제 노트북(notebook). 을 참조하십시오. 커스텀 모델을 로드하려면,
preproc_func(img)와 같은 전처리 함수를 작성해야 하며, 이 함수는 입력 tensor가 로드된 네트워크에 적합한 형식으로 되어 있는지 확인해줍니다.
또한,
사전 학습된 모델(pretrained_model)을 문자열 인수로 전달할 수 있습니다. 이는 예측을 수행할 CNN 모델을 지정하며, 해당 모델은 여기 나열된 모델 중 하나이어야 합니다. 명령어는 다음과 같습니다:predictor = PatchPredictor(pretrained_model='resnet18-kather100k', pretrained_weights=weights_path, batch_size=32).pretrained_weights:사전 학습된 모델(pretrained_model)을 사용할 때, 해당 모델의 사전 학습된 가중치도 기본적으로 다운로드 됩니다. 기본으로 제공되는 가중치를 덮어쓰고 자신만의 가중치를 사용하려면pretrained_weight인수를 통해 가중치를 지정할 수 있습니다.batch_size: 모델에 한 번에 입력되는 이미지의 개수를 지정합니다. 이 값이 클수록 더 많은 (GPU)메모리 용량이 필요합니다.
# TIAToolbox에서 사전 학습된 PyTorch 모델 가져오기
predictor = PatchPredictor(pretrained_model='resnet18-kather100k', batch_size=32)
# 사용자는 아래 스크립트를 통해 원하는 PyTorch 모델 아키텍처를 불러올 수 있습니다.
model = vanilla.CNNModel(backbone="resnet18", num_classes=9) # torchvision.models.resnet18에서 모델 불러오기
model.load_state_dict(torch.load(weights_path, map_location="cpu", weights_only=True), strict=True)
def preproc_func(img):
img = PIL.Image.fromarray(img)
img = transforms.ToTensor()(img)
return img.permute(1, 2, 0)
model.preproc_func = preproc_func
predictor = PatchPredictor(model=model, batch_size=32)
패치 라벨 예측하기#
예측기(predictor) 객체를 생성한 후 patch 모드를 사용하여 predict 메소드를 호출합니다.
그런 다음, 분류 정확도와 오차 행렬(confusion matrix)을 계산합니다.
with suppress_console_output():
output = predictor.predict(imgs=patch_list, mode="patch", on_gpu=ON_GPU)
acc = accuracy_score(label_list, output["predictions"])
logger.info("Classification accuracy: %f", acc)
# 패치 분류 결과를 위한 오차 행렬(confusion_matrix) 생성 및 시각화
conf = confusion_matrix(label_list, output["predictions"], normalize="true")
df_cm = pd.DataFrame(conf, index=class_names, columns=class_names)
df_cm
|2023-11-14|13:16:03.215| [INFO] Classification accuracy: 0.993000
전체 슬라이드(whole slide)에 대한 패치 라벨 예측#
IOPatchPredictorConfig 클래스를 소개합니다. 이 클래스는 모델 예측 엔진을
위한 이미지 읽기 및 예측 결과 쓰기 구성 설정을 지정합니다.
이 설정은 분류기(classifier)에게 WSI 피라미드의 어느 레벨을 읽고,
데이터를 처리하며, 출력을 생성해야 하는지 알려주는 데
필수적입니다.
IOPatchPredictorConfig 의 매개변수는 다음과 같이 정의됩니다.
input_resolutions: 입력의 해상도를 지정하는 딕셔너리 형태의 리스트로, 각 입력의 해상도를 설정합니다. 리스트의 요소는model.forward()의 순서와 같아야합니다. 만약 모델이 하나의 입력만 받는 경우, 하나의 딕셔너리로'units'와'resolution'을 지정하면 됩니다. TIAToolbox는 하나 이상의 입력을 받는 모델을 지원하므로, 여러 입력을 사용하는 경우에도 문제없이 사용할 수 있습니다. 유닛(units)과 해상도(resolution)에 대한 자세한 내용은 TIAToolbox 문서 를 참고하십시오.patch_input_shape: 가장 큰 입력의 크기를 (높이, 너비) 형식으로 지정합니다.stride_shape: 연속된 두 패치 사이의 간격(단계) 크기를 지정하며, 패치 추출 과정에서 사용됩니다. 사용자가stride_shape를patch_input_shape와 동일하게 설정하면, 패치들이 중첩없이 추출되고, 처리됩니다.
wsi_ioconfig = IOPatchPredictorConfig(
input_resolutions=[{"units": "mpp", "resolution": 0.5}],
patch_input_shape=[224, 224],
stride_shape=[224, 224],
)
predict 메소드는 입력 패치에 CNN을 적용하여 결과를 얻습니다.
다음은 해당 메소드의 인수와 설명입니다:
mode: 처리할 입력의 유형을 지정합니다. 응용 프로그램에 따라patch,tile또는wsi중에서 선택합니다.imgs: 입력 파일들의 경로 리스트로, 입력 타일(input tiles) 또는 WSIs 경로의 목록이어야 합니다.return_probabilities: 입력 패치의 예측된 라벨과 함께 클래스별 확률을 얻으려면 True 로 설정합니다.tile또는wsi모드에서 예측 결과를 병합하여 예측 맵을 생성하려면return_probabilities=True로 설정할 수 있습니다.ioconfig:IOPatchPredictorConfig클래스를 사용하여 IO 구성 정보를 설정합니다.resolution과unit(아래에 표시되지 않음): 추출할 패치의 WSI 레벨 또는 마이크론당 픽셀 해상도를 지정합니다. 이는ioconfig대신 사용할 수 있습니다. 여기서 WSI 레벨은'baseline'으로 지정하며, 이는 일반적으로 레벨 0에 이 경우 이미지는 하나의 레벨만 가지고 있습니다. 더 자세한 내용은 문서 에서 확인할 수 있습니다.masks:imgs리스트에 있는 WSI의 마스크 경로 리스트입니다. 이 마스크는 원본 WSI에서 패치를 추출하고자 하는 영역을 지정합니다. 특정 WSI의 마스크가None으로 지정되면, 해당 WSI의 모든 패치(배경 영역 포함)에 대한 라벨이 예측됩니다. 이는 불필요한 계산을 유발할 수 있습니다.merge_predictions: 패치 분류 결과를 2D 맵으로 생성해야하는 경우True로 설정할 수 있습니다. 그러나 큰 WSI의 경우 많은 메모리가 필요할 수 있습니다. 대안적인 해결책으로는merge_predictions=False로(기본값) 설정하여, 추후에merge_predictions함수를 사용해 2D 예측 맵을 생성하는 방법을 사용할 수 있습니다.
큰 WSI를 사용하고 있기 때문에 패치 추출 및 예측 과정에 시간이 다소 걸릴 수 있습니다.
(만약 Cuda가 활성화된 GPU를 사용할 수 있다면
ON_GPU=True 로 설정하여 PyTorch와 Cuda를 활용하는 것이 좋습니다).
with suppress_console_output():
wsi_output = predictor.predict(
imgs=[wsi_path],
masks=None,
mode="wsi",
merge_predictions=False,
ioconfig=wsi_ioconfig,
return_probabilities=True,
save_dir=global_save_dir / "wsi_predictions",
on_gpu=ON_GPU,
)
wsi_output 을 시각화하여 예측 모델이 전체 슬라이드 이미지(WSI)에서
어떻게 작동하는지 확인할 수 있습니다. 먼저 패치 예측 결과를 병합한 후,
이를 원본 이미지 위에 오버레이로 시각화해야 합니다. 이전과 마찬가지로
merge_predictions 메소드를 사용하여 패치 예측을 병합합니다.
이때, 1.25x 만큼 확대된 예측 맵을 생성하기 위해
resolution=1.25, units='power' 로 매개변수를 설정합니다.
만약 더 높은/낮은 해상도 (더 큰/작은) 예측 맵을 원한다면,
이 매개변수를 적절히 변경해야 합니다.
예측이 병합되면 overlay_patch_prediction 함수를 사용하여
예측 맵을 WSI 썸네일에 오버레이합니다. 이때 사용된 해상도는
예측 병합에 사용된 해상도와
일치해야합니다.
overview_resolution = (
4 # 패치 예측을 병합하고 시각화하는 해상도 설정
)
# '해상도' 매개변수의 단위 설정. "power", "level", "mpp", 또는 "baseline" 중에서 선택 가능
overview_unit = "mpp"
wsi = WSIReader.open(wsi_path)
wsi_overview = wsi.slide_thumbnail(resolution=overview_resolution, units=overview_unit)
plt.figure(), plt.imshow(wsi_overview)
plt.axis("off")
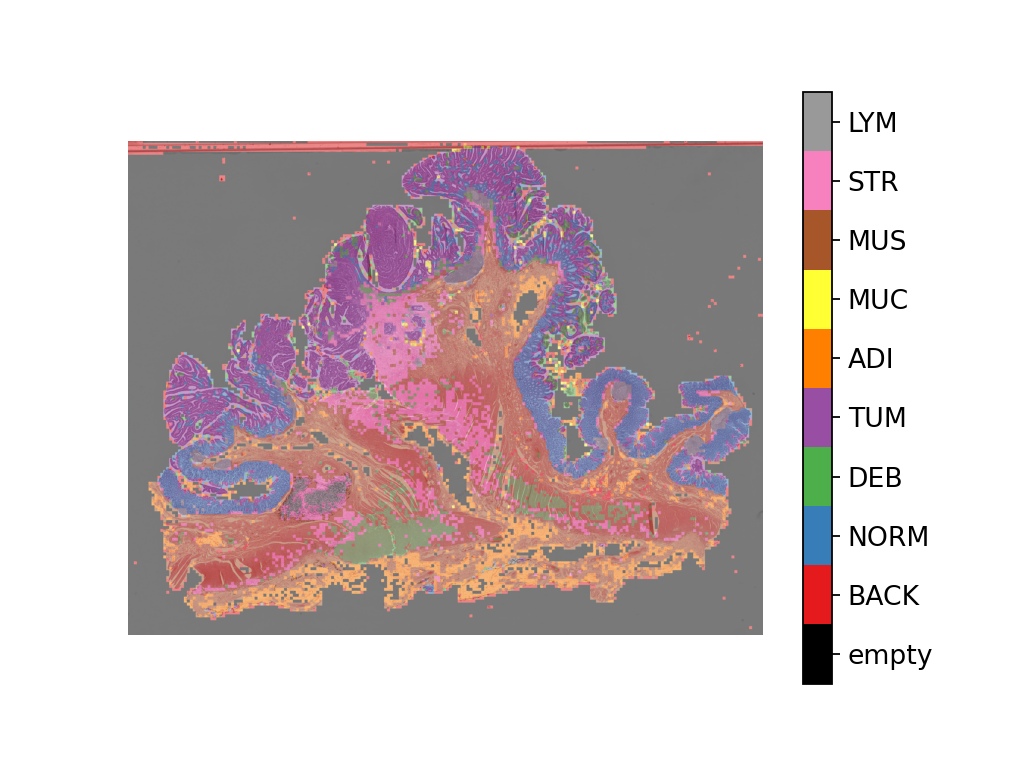
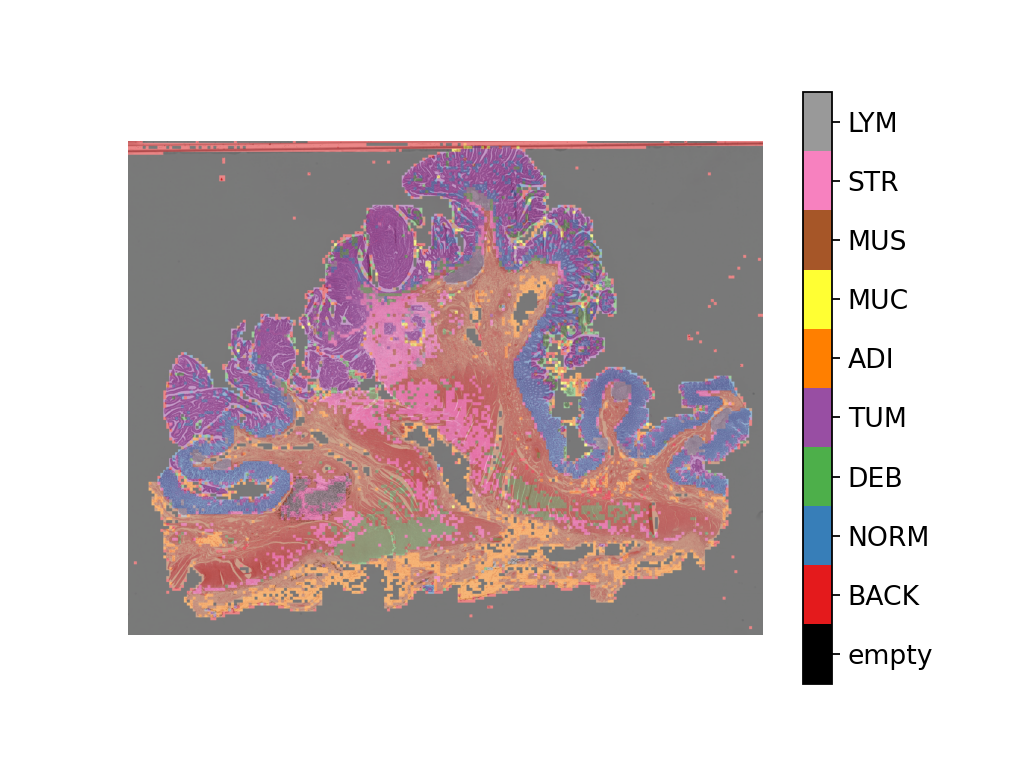
예측 맵을 이 이미지에 오버레이한 결과는 다음과 같습니다:
# 전체 슬라이드 이미지(Whole slide image)의 패치 레벨 예측 시각화
# 먼저 라벨 색상의 매핑 설정
label_color_dict = {}
label_color_dict[0] = ("empty", (0, 0, 0))
colors = cm.get_cmap("Set1").colors
for class_name, label in label_dict.items():
label_color_dict[label + 1] = (class_name, 255 * np.array(colors[label]))
pred_map = predictor.merge_predictions(
wsi_path,
wsi_output[0],
resolution=overview_resolution,
units=overview_unit,
)
overlay = overlay_prediction_mask(
wsi_overview,
pred_map,
alpha=0.5,
label_info=label_color_dict,
return_ax=True,
)
plt.show()
병리학에 특화된 모델을 사용한 특징 추출#
이 부분에서는 TIAToolbox 외부에 존재하는 사전 학습된 PyTorch 모델에서 특징을 추출하는 방법을 TIAToolbox에서 제공하는 WSI 추론 엔진을 사용하여 보여줍니다. 이를 설명하기 위해, HistoEncoder라는 병리학적 이미지에 특화된 모델을 사용할 것입니다. HistoEncoder는 조직학 이미지에서 특징을 추출하도록 자가 지도 학습 방식(self-supervised) 으로 학습되었습니다. 이 모델은 다음에서 사용할 수 있습니다:
‘HistoEncoder: 디지털 병리학을 위한 기본 모델’ (jopo666/HistoEncoder) 헬싱키 대학교 Pohjonen, Joona 팀.
특징 맵의 umap 차원 축소를 3D(RGB)로 시각화하여, 위에서 언급한 여러 조직 유형 간의 차이를 특징들이 어떻게 포착하는지 보여줄 것입니다.
# 추가 module 가져오기
import histoencoder.functional as F
import torch.nn as nn
from tiatoolbox.models.engine.semantic_segmentor import DeepFeatureExtractor, IOSegmentorConfig
from tiatoolbox.models.models_abc import ModelABC
import umap
TIAToolbox는 PyTorch의 nn.Module 을 상속하는 ModelABC 클래스를 정의하며, 이는 TIAToolbox의 추론 엔진에서 사용될 모델의 구조를 규정합니다. 하지만 histoencoder 모델은 이 구조를 따르지 않기 때문에, TIAToolbox 엔진이 기대하는 출력과 메소드를 제공하는 클래스로 HistoEncoder를 래핑(wrap)해야 합니다.
class HistoEncWrapper(ModelABC):
"""tiatoolbox의 ModelABC 인터페이스에 맞춘 HistoEncW모델의 레퍼 생성"""
def __init__(self: HistoEncWrapper, encoder) -> None:
super().__init__()
self.feat_extract = encoder
def forward(self: HistoEncWrapper, imgs: torch.Tensor) -> torch.Tensor:
"""입력 데이터를 모델을 통해 전달
Args:
imgs (torch.Tensor):
Model input.
"""
out = F.extract_features(self.feat_extract, imgs, num_blocks=2, avg_pool=True)
return out
@staticmethod
def infer_batch(
model: nn.Module,
batch_data: torch.Tensor,
*,
on_gpu: bool,
) -> list[np.ndarray]:
"""입력 배치에 대한 추론 실행
순방향 연산과 입출력 집계를 위한 로직이 포함되어 있습니다.
Args:
model (nn.Module):
정의된 PyTorch 모델.
batch_data (torch.Tensor):
`torch.utils.data.DataLoader`에
의해 생성된 데이터의 배치(batch).
on_gpu (bool):
추론 연산을 GPU에서 할 것인지.
"""
img_patches_device = batch_data.to('cuda') if on_gpu else batch_data
model.eval()
# 기울기를 계산하지 않음(훈련이 아님)
with torch.inference_mode():
output = model(img_patches_device)
return [output.cpu().numpy()]
이제 래퍼(wrapper)를 만들었으니, 특징 추출 모델을 생성하고 DeepFeatureExtractor 를 인스턴스화 하여 이 모델을 WSI에 사용할 수 있게 합니다. 이전에 사용했던 동일한 WSI를 사용하지만, 이번에는 각 패치에 대한 라벨을 예측하는 대신 HistoEncoder 모델을 사용하여, WSI의 패치에서 특징을 추출할 것입니다.
# 모델 만들기
encoder = F.create_encoder("prostate_medium")
model = HistoEncWrapper(encoder)
# 전처리 함수 설정
norm=transforms.Normalize(mean=[0.662, 0.446, 0.605],std=[0.169, 0.190, 0.155])
trans = [
transforms.ToTensor(),
norm,
]
model.preproc_func = transforms.Compose(trans)
wsi_ioconfig = IOSegmentorConfig(
input_resolutions=[{"units": "mpp", "resolution": 0.5}],
patch_input_shape=[224, 224],
output_resolutions=[{"units": "mpp", "resolution": 0.5}],
patch_output_shape=[224, 224],
stride_shape=[224, 224],
)
DeepFeatureExtractor 를 생성할 때
auto_generate_mask=True 인수를 전달할 것입니다. 이는 otsu 임계값
알고리즘을 사용하여 자동으로 조직 영역의 마스크를 생성하여, 추출기가
조직이 포함된 패치에만 처리하도록 합니다.
# 특징 추출기 생성 및 WSI에서 실행하기
extractor = DeepFeatureExtractor(model=model, auto_generate_mask=True, batch_size=32, num_loader_workers=4, num_postproc_workers=4)
with suppress_console_output():
out = extractor.predict(imgs=[wsi_path], mode="wsi", ioconfig=wsi_ioconfig, save_dir=global_save_dir / "wsi_features",)
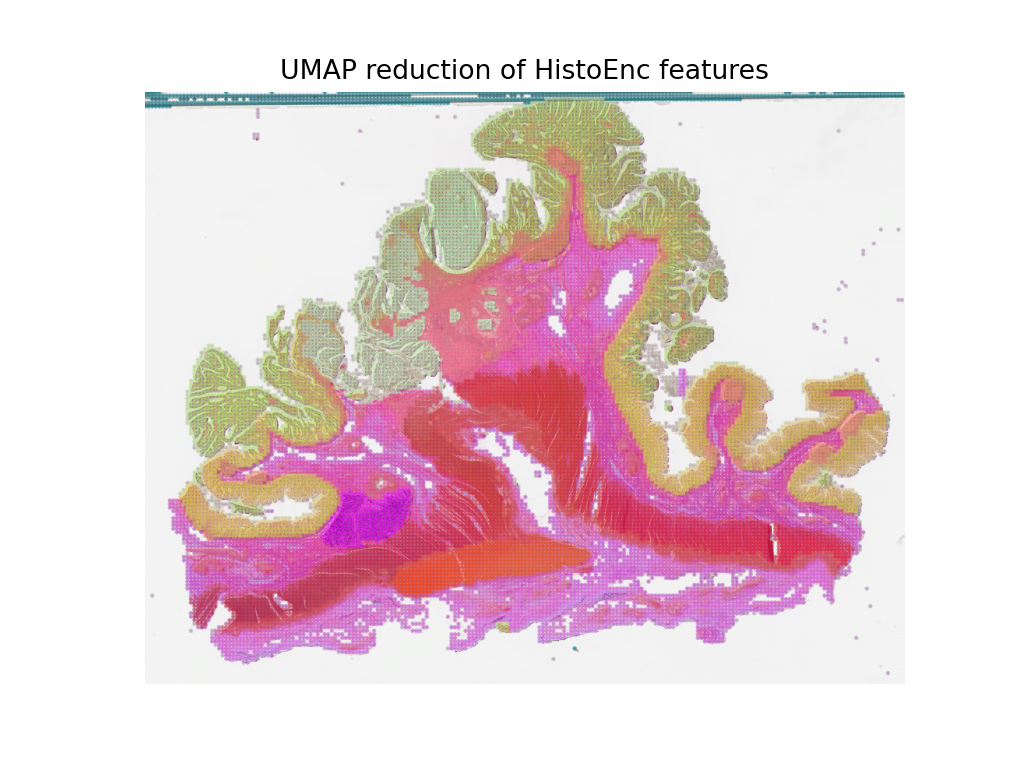
이러한 특징들은 다운스트림 모델을 훈련하는 데 사용할 수 있지만, 여기서는 특징이 무엇을 나타내는지 직관적으로 이해하기 위해 UMAP 차원 축소를 사용하여 특징을 RGB 공간에서 시각화할 것입니다. 유사한 색상으로 라벨링된 포인트들은 비슷한 특징을 가지고 있어야 하므로, UMAP 축소 결과를 WSI 썸네일에 오버레이하여 특징들이 서로 다른 조직 영역으로 자연스럽게 분리되는지 확인할 수 있습니다. 이후, 위에서 생성한 패치 수준의 예측 맵과 함께 이를 시각화하여, 특징과 패치 수준 예측 간의 차이를 비교해보겠습니다.
# 먼저, UMAP 축소 계산을 위한 함수 정의
def umap_reducer(x, dims=3, nns=10):
"""입력 데이터의 UMAP 축소"""
reducer = umap.UMAP(n_neighbors=nns, n_components=dims, metric="manhattan", spread=0.5, random_state=2)
reduced = reducer.fit_transform(x)
reduced -= reduced.min(axis=0)
reduced /= reduced.max(axis=0)
return reduced
# 특징 추출기에서 출력된 특징 불러오기
pos = np.load(global_save_dir / "wsi_features" / "0.position.npy")
feats = np.load(global_save_dir / "wsi_features" / "0.features.0.npy")
pos = pos / 8 # 0.5mpp에서 특징을 추출하고, 4mpp에서 썸네일에 오버레이하기
# 특징을 3차원(RGB) 공간으로 축소하기
reduced = umap_reducer(feats)
# 분류기의 예측 맵을 다시 그리기
overlay = overlay_prediction_mask(
wsi_overview,
pred_map,
alpha=0.5,
label_info=label_color_dict,
return_ax=True,
)
# 특징 맵 축소를 시각화하기
plt.figure()
plt.imshow(wsi_overview)
plt.scatter(pos[:,0], pos[:,1], c=reduced, s=1, alpha=0.5)
plt.axis("off")
plt.title("UMAP reduction of HistoEnc features")
plt.show()
패치 수준 예측기(patch-level predictor)에서 생성된 예측 맵과 자가 지도 학습(self-supervised) 인코더를 통해 특징을 추출한 특징 맵이 WSI에서 조직 유형에 대한 유사한 정보를 포착하고 있음을 확인할 수 있습니다. 이는 모델이 예상대로 작동하고 있음을 확인할 수 있는 좋은 검증 방법입니다. 또한, HistoEncoder 모델이 추출한 특징들이 조직 유형 간의 차이를 잘 포착하고 있으며, 따라서 이 특징들이 조직학적으로 중요한 정보를 인코딩하고 있음을 보여줍니다.
앞으로 해야할 것#
이 노트북에서는 PatchPredictor 와
DeepFeatureExtractor 클래스의 predict 메소드를 사용하여
큰 타일(tiles)과 WSI의 패치에 대해 라벨을 예측하거나 특징을 추출하는 방법을 보여줍니다.
또한, 패치 예측 결과를 병합하고 입력 이미지/WSI에 예측 맵을
오버레이로 시각화하는 merge_predictions 와 overlay_prediction_mask
보조 함수도 소개합니다.
모든 과정은 TIAToolbox 내에서 이루어지며, 예제 코드를 따라 쉽게 구성할 수 있습니다. 입력값과 옵션을 올바르게 설정하는 것을 꼭 확인하세요. 또한 predict 함수의 매개변수를 변경했을 때 예측 결과에 미치는 영향을 탐구하는 것도 권장합니다. TIAToolbox 프레임워크에서 제공하는 커뮤니티의 모델 또는 사용자 정의 사전 학습된 모델을 사용하여, TIAToolbox 모델 클래스에 정의되지 않은 구조의 모델이라도 대형 WSI에 대해 추론을 수행하는 방법을 시연했습니다.
다음 자료를 통해 더 많은 내용을 배울 수 있습니다: